客製化服務, GeneDireX 客製化服務, PRIME Gene Synthesis
基因合成基本觀念
基因合成是一個具有革命性的技術,與在細胞中發生的DNA複製及經由PCR放大不同,基因合成不需要DNA模板就能合成目標基因序列,幾乎任何序列都可以合成,包括不存在於自然界中的序列,科學家不再侷限於傳統分生克隆,可以設計或是重新編碼整個基因組來合成。
基因合成的原理
一般基因合成是由一個鹼基一個鹼基,經由化學合成方式逐步地添加而形成一條DNA鏈,這方法叫做亞磷醯胺合成法(phosphoramidite chemistry),是常見DNA固相的合成方式,包含四個步驟:
1. 去保護基(deprotection),將5’羥基(hydroxy group)上的保護基dimethoxytrityl(DMT)去除
2. 耦合反應(coupling),第2個鹼基(帶有DMT)與第1個鹼基形成亞磷酸三酯鍵(phosphite triester)耦合在一起
3. 帶帽反應(capping),少數未參與耦合反應的羥基醯化(acylation)作用,防止其後繼續發生反應
4. 氧化反應(oxidation),耦合反應形成的亞磷酸三酯鍵並不穩定,再進行下一個循環前必須使其氧化,轉為較穩定的磷酸三酯鍵(phosphate triester)

圖一、基因合成: 亞磷醯胺合成法
重複此四個步驟直到形成一條DNA鏈,但因限制於化學反應的效率,能合成的基因片段無法太長,較不適合大規模合成。
新型合成技術
新型的合成技術是結合矩陣式及亞磷醯胺合成法來進行基因合成,可用於高通量的序列合成,目前仍在持續發展矩陣式的相關技術,未來能提供更強大且高品質的基因序列合成方法,分為四個步驟:
- 在矩陣中合成寡核甘酸(oligonucleotide)序列
- 次世代定序來鑑定在矩陣中合成的序列
- 從合成片段的群集(pool)中選出要放大的片段
- 放到子矩陣中放大並組裝成基因片段來完成基因的構築
針對高難度或特長合成序列無法一次完成,需經由基因片段組裝,常用的有三種:
- 接合連鎖反應(ligation chain reaction, LCR),相鄰單鏈寡核甘酸設計成有重疊且互補的片段,將這些寡核甘酸混合在一起,經由升降溫解鏈黏合使其成為雙股DNA片段,重複步驟直到有全鏈的基因序列,最後用PCR擴增
- 相鄰的單鏈寡核甘酸在二端有一部分是重疊的互補序列,利用PCR反應形成雙股DNA,此法稱聚合酶連鎖組裝(polymerase cycle assembly, PCA)
- 接合反應(ligation)與PCA結合,經由PCA反應組裝好的基因片段,再經接合反應直接接到載體上
針對取得多條基因序列的需求,相較於傳統多次克隆的方式,基因合成更可節省操作時間及成本,而且一般傳統做出來的質體常常蛋白表現量不高,難以進行後續實驗,不過基因合成可以透過密碼子的優化(codon optimization)來提高目標蛋白質的表現量。 基因合成流程,如圖二,將合成好的基因序列再經由克隆技術接合到載體上,可以維持插入序列在操作及保存期間較不會降解,定序驗證插入序列的正確性 。
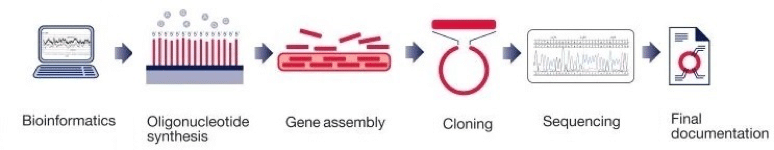
圖二、基因合成流程(註10)
傳統克隆作法:
- 從細胞或組織中抽取RNA,反轉錄成cDNA
- 設計引子,利用PCR放大基因片段
- 純化PCR產物
- 經由限制酶剪切接合上載體
- 完成一個質體,送入細菌中後挑出菌落
- 定序確認序列
傳統克隆方式取得序列的瓶頸:
- 抽出的RNA量小且不穩定
- 需要調整PCR的最適化條件
- 連續重複序列(tandem repeat sequence)及長片段重覆鹼基增加PCR難度
- 純化PCR產物時,損失過多的產物
- 接合作用效率低
- 多次PCR產生突變
- 難以完成密碼子優化與致突變作用序列
委託服務要點與注意事項
- 填寫基因名稱
- 基因序列或蛋白質序列
- 限制酶切位的選擇: 接進載體裡需要序列兩端加上限制酶切位序列,並且不 含在目標基因序列中
- 序列優化與否: 依照實驗目的不同選擇,序列優化可提高蛋白表現以及序列中GC含量分布均勻
- 序列優化要避免的限制酶切位: 通常會避免要接到載體上的兩端限制酶
- 抗生素選擇: ampicillin及kanamycin
- 載體選擇: 穿梭載體(shuttle vector)或表現載體(expression vector)
密碼子優化(codon optimization)的重要性
研究基因功能、蛋白質結構、酵素動力或是藥物研發時,時常需要將目標蛋白大量表現,利用在宿主細胞中來表達目標蛋白的基因,而這宿主細胞與原先序列可能是不同物種,此時便會發生這基因在宿主細胞中所表達的蛋白質過低,也會因產生的蛋白過低而造成蛋白質不當的摺疊,這是因為不同物種間使用的遺傳密碼子偏好所造成的,如表一,而序列優化是指現在利用生資系統,在不改變最後轉譯出胺基酸序列的原則下,改變遺傳密碼子以突破不同物種間密碼子使用偏好的問題,這不僅提高了蛋白的表現,也提高了所產出來蛋白的正確性。
| Alanine | Human | E. coli |
| GCG | 7.6% | 30.1% |
| Proline | Human | E. coli |
| CCC | 20.0% | 5.4% |
| CCG | 7.0% | 20.9% |
表一、同個胺基酸在不同物種間遺傳密碼子使用的偏好
現今生產蛋白有許多不同的系統,哺乳類細胞(例如: CHO)、昆蟲細胞(例如: sf9)、酵母菌(例如:pichia)以及細菌(例如:E. coli),需依照所選擇表達系統的物種來執行序列的優化。 序列優化目的不只有提高蛋白表現及正確性,有些序列結構上較具合成難度,例如:過高或過低的GC含量,有複雜的二級結構及有polyA的延伸,這些都是重要的因素需要將原序列優化,使GC含量分布均勻,避免二級結構等來讓基因順利的表達。
Reference
- Siying Ma, Nicholas Tang, and Jingdong Tian DNA Synthesis, Assembly and Applications in Synthetic Biology Curr Opin Chem Biol. 2012 August
- Randall A. Hughes and Andrew D. Ellington Synthetic DNA Synthesis and Assembly: Putting the Synthetic in Synthetic Biology Cold Spring Harb Perspect Biol 2017
- Jingdong Tian, Kuosheng Ma and Ishtiaq Saaem Advancing high-throughput gene synthesis technology Molecular BioSystems 2009
- Robert M. Nowak et al. DNASynth: A Computer Program for Assembly of Artificial Gene Parts in Decreasing Temperature BioMed Research International 2014
- Jiayuan Quan et al. Parallel on-chip gene synthesis and application to optimization of protein expression Nature biotechnology 2011
- Ai-Sheng Xiong et al. Non-polymerase-cycling-assembly-based chemical gene synthesis: strategies, methods, and progress Biotechnology Advances 2008
- Annabel HA Parret, Hu¨ seyin Besir and Rob Meijers Critical reflections on synthetic gene design for recombinant protein expression Structural Biology 2016
- Joshua B. Plotkin and Grzegorz Kudla Synonymous but not the same: the causes and consequences of codon bias Nat Rev Genet. 2011
- Stanley N. Cohen DNA cloning: A personal view after 40 years PNAS 2013
- https://www.thermofisher.com/us/en/home/life-science/cloning/gene-synthesis/geneart-gene-synthesis.html
